近日���,真邁生物在BMC bioinformatics上發(fā)表了題為“Systematic and benchmarking studies of pipelines for mammal WGBS data in the novel NGS platform”的研究成果��。該研究在真邁生物GenoLab M高通量測序平臺和Illumina的NovaSeq 6000平臺進行全基因組甲基化測序,比較了兩個平臺對小鼠肝臟細胞系NCTC1469和人胚胎腎細胞系293(HEK293)的全基因組甲基化圖譜的差異與影響��,并探究了GenoLab M平臺對于目前5款常用甲基化分析流程(BSMAP�����,Bismark��,BS Seeker2�,BatMeth2,BSBolt)的偏好性�。
背景介紹
全基因組甲基化測序(Whole?Genome?Bisulfite?Sequencing,WGBS)主要是通過重亞硫酸鹽將基因組序列中沒有發(fā)生甲基化的胞嘧啶(Cytosine--C)轉(zhuǎn)化為尿嘧啶����,再通過高通量測序平臺,對胞嘧啶的甲基化水平進行檢測��,精確解析每一個胞嘧啶(C)的甲基化狀態(tài)����,從而構(gòu)建全基因組甲基化圖譜,深入探究甲基化胞嘧啶在人類疾病��、生長發(fā)育過程中的重要作用���。
01項目研究概要
本研究采用小鼠肝臟細胞系和人胚胎腎細胞系樣本�,各有2個生物學(xué)重復(fù)�����,構(gòu)建完甲基化文庫后���,文庫一分為二�����,分別在GenoLab M平臺和NovaSeq 6000平臺測序�����,共獲得8個下機數(shù)據(jù)�。隨后完成全基因組甲基化分析,解析不同平臺的甲基化圖譜分布的影響和差異���,以及GenoLab M平臺數(shù)據(jù)的分析軟件偏好���。
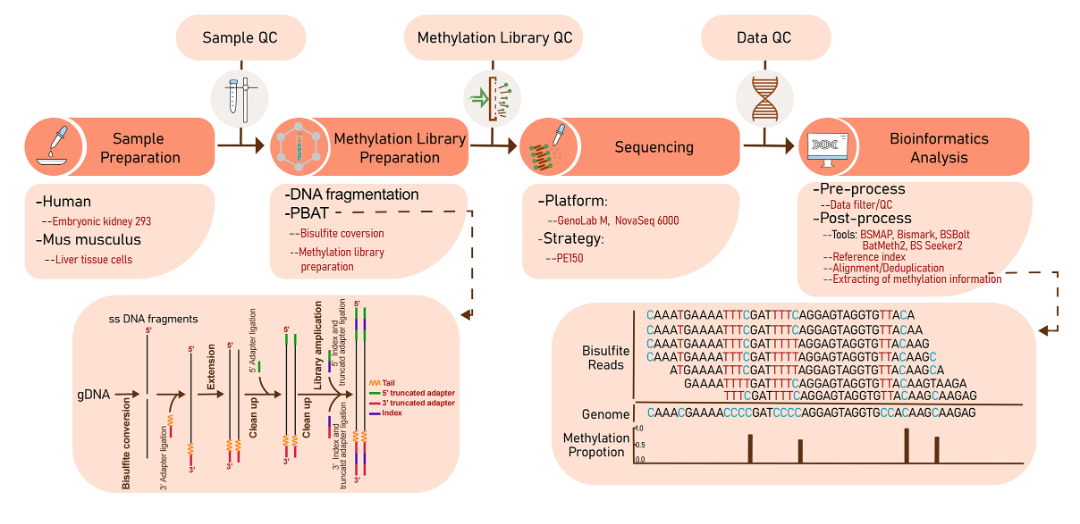
圖1 關(guān)鍵的干����、濕實驗流程圖
02數(shù)據(jù)質(zhì)控-GenoLab M平臺數(shù)據(jù)質(zhì)量更好
兩個平臺下機序列(reads)開頭和結(jié)尾的幾個堿基質(zhì)量較低���,這會影響后續(xù)序列在全基因組上的比對準確性�����。因此����,我們比較了截取不同長度堿基后序列的比對情況��。發(fā)現(xiàn)�����,將read1序列的結(jié)尾和read2序列的開頭分別截取掉10個堿基���,可以有效提升全基因組范圍內(nèi)的唯一比對準確率�����,參見圖2 a和c圖���。基于上述處理后���,我們發(fā)現(xiàn)���,同等下機數(shù)據(jù)情況下�����,GenoLab M平臺的有效數(shù)據(jù)產(chǎn)出更多��。
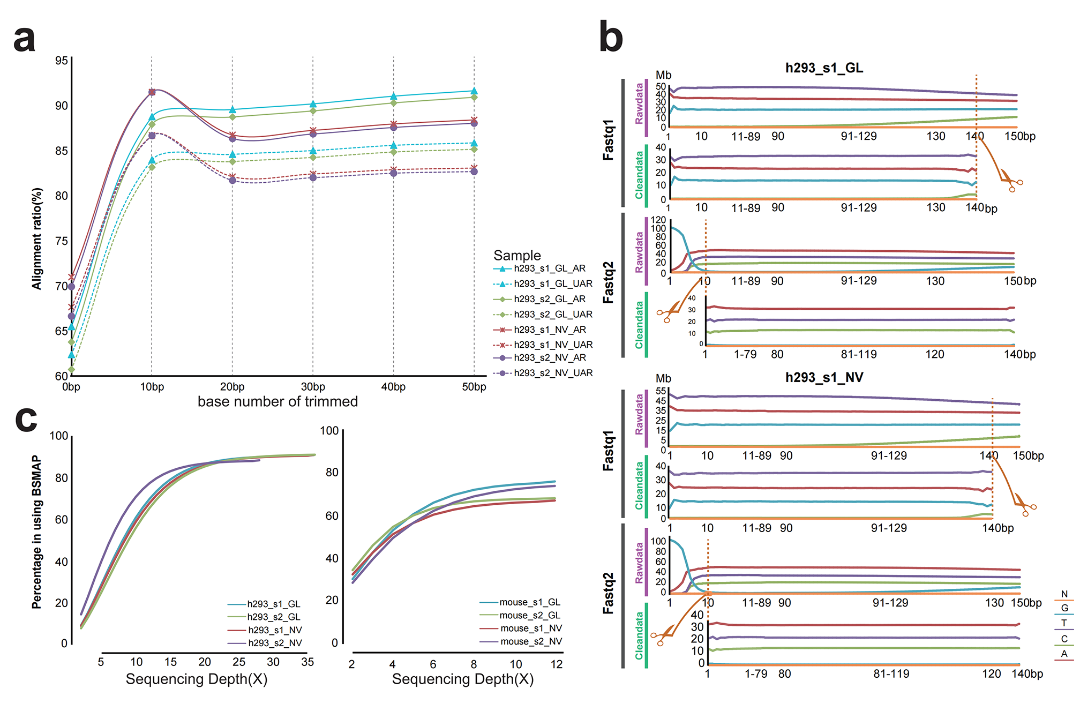
圖2?過濾步驟中reads的最優(yōu)base剪切數(shù)
03甲基轉(zhuǎn)化率平臺間差異表現(xiàn)和分析軟件偏好類似
隨后����,我們分別使用了五個常用的分析軟件進行全基因組的比對分析和甲基化C分布檢測的平行比較�����。結(jié)果發(fā)現(xiàn)�,GenoLab M平臺數(shù)據(jù)的重復(fù)序列相對NovaSeq 6000更低(人3.05% VS 12.38%,鼠2.82% VS 17.24%)�����。兩個測序平臺的數(shù)據(jù)使用BSBolt軟件均可獲得最高的唯一比對率(人94%,小鼠91%)��;其次是BetMath2軟件(人91%�,小鼠86%)。兩個測序平臺產(chǎn)出數(shù)據(jù)使用BS Seeker2比對獲取到的唯一比對率最低��。五個軟件檢測到的甲基化C占比均為70~80%范圍內(nèi)�。
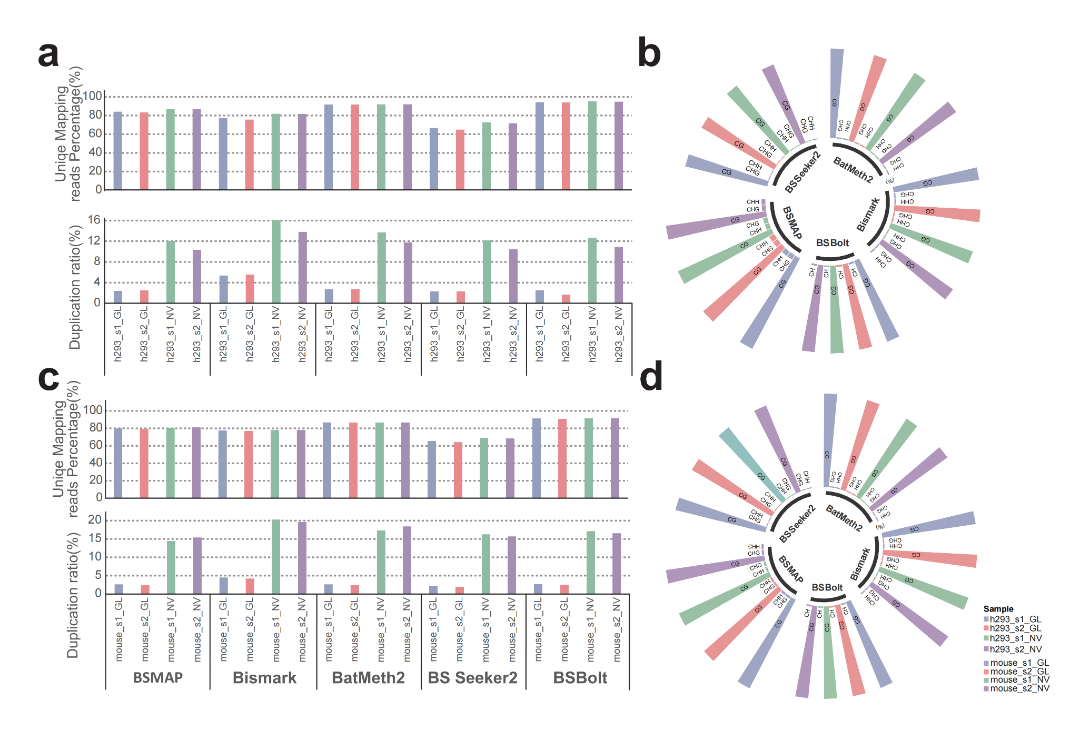
圖3?不同軟件檢測到的比對率和甲基化Cs(CG�����、CHG����、CHH)的占比
04全基因組甲基化圖譜一致性評估
使用五個軟件對兩個平臺產(chǎn)出的數(shù)據(jù)進行分析,我們發(fā)現(xiàn)���,所有軟件獲取的全基因組范圍內(nèi)CG序列甲基化率分布圖譜(mCGs)均具有較高的一致性���,但是CHG和CHH序列甲基化率分布圖譜差異較大�。通過檢測不同生物學(xué)重復(fù)樣本所有的甲基化C位點(mCs)和CG序列甲基化的C位點(mCGs)��,我們發(fā)現(xiàn)����,相對于NovaSeq 6000平臺,GenoLab M平臺數(shù)據(jù)具有更多的mCs和mCGs位點����,且在不同生物學(xué)重復(fù)樣本中,這些mCs和mCGs具有更高的一致性����。
比較五個分析軟件所能檢測到的全基因組C,我們發(fā)現(xiàn)�,在生物學(xué)重復(fù)樣本中,BSMAP���、BS Seeker2和Bismark獲取到的mCGs的結(jié)果一致性更高�����,且結(jié)果可以相互驗證���。綜合比較���,BSMAP軟件獲取的結(jié)果相對來說最好,且耗時也相對最少�����。
表格1 五個分析軟件的配置和分析HEK293樣本數(shù)據(jù)的時間消耗

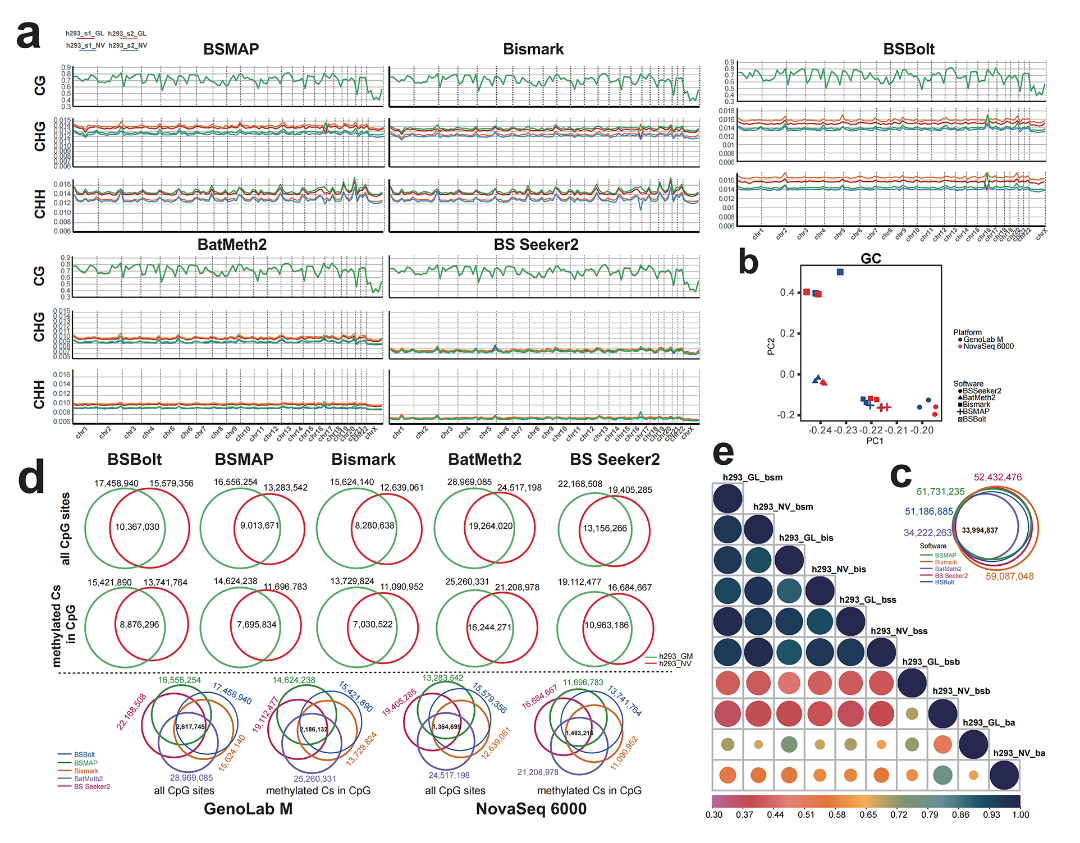
圖4 兩個平臺產(chǎn)出數(shù)據(jù)在不同軟件檢測出的全基因組圖譜的比較
05mCGs圖譜與早期分析的mCGs圖譜的比較
基于BSMAP軟件獲取的全基因組甲基化圖譜在基因附近的分布分析�����,我們發(fā)現(xiàn)��,不管是在人細胞系中���,還是在小鼠細胞系中�����,兩個平臺的結(jié)果一致性均較高���。通過與早期已發(fā)表的HEK293細胞系的全基因組甲基化圖譜結(jié)果比較,并重點關(guān)注了兩個基因(GNB1和NSUN2)中甲基化位點的分布情況��,我們發(fā)現(xiàn)兩個平臺數(shù)據(jù)集和早期的三個數(shù)據(jù)集中這兩個基因的甲基化圖譜一致性均較高�����。
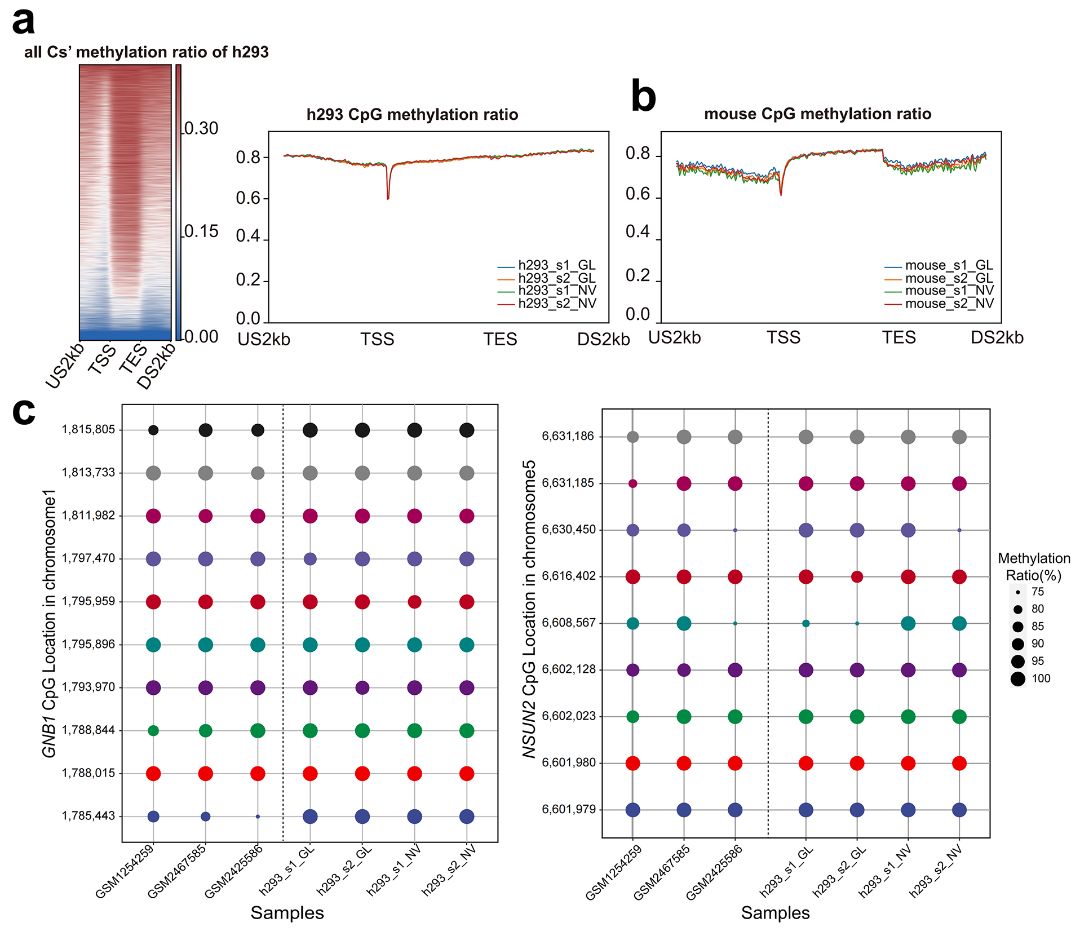
圖5?相關(guān)基因甲基化圖譜的一致性比較
結(jié)論
基于上述全面的分析�,我們得出如下結(jié)論:
1?、GenoLab M與NovaSeq 6000兩個平臺產(chǎn)出的甲基化數(shù)據(jù)通過有效截斷過濾����,可顯著提升序列比對準確性;
2 ����、GenoLab M平臺測序數(shù)據(jù)的重復(fù)序列更低���,有效序列更多;
3�����、?GenoLab M平臺數(shù)據(jù)包含更多有效的mCs和mCGs位點�,且在人和鼠生物學(xué)重復(fù)樣本中,其結(jié)果一致性更高����;
4 、五個常用甲基化分析軟件中���,BSMAP的結(jié)果最好,相對耗時更短�����。
討論
甲基化測序技術(shù)作為研究生命過程中基因調(diào)控的重要工具���,在細胞分化和疾病進展方面得到了廣泛的應(yīng)用�,臨床檢測方面的應(yīng)用也越來越多����。例如:腫瘤早篩-進展監(jiān)測�、精準治療選擇�����、微小殘留監(jiān)測��、預(yù)后-復(fù)發(fā)檢測等�����。目前��,有研究發(fā)現(xiàn)���,含有游離核酸的體液樣本中可能含有腫瘤特異的DNA甲基化信號�,它們可能是潛在的生物標志物����。GenoLab M平臺在本次WGBS全面分析中表現(xiàn)出測序質(zhì)量更穩(wěn)定、更高���,數(shù)據(jù)覆蓋度更深����,重復(fù)率更低,甲基化一致性水平更高�,準確率更好等性能,能滿足更多的甲基化測序項目需求���。